ARN -la polymérase est une enzyme qui synthétise les molécules d'ARN. Au sens étroit, l'ARN polymérase est généralement appelée ARN polymérases ADN-dépendantes qui synthétisent des molécules d'ARN sur une matrice d'ADN, c'est-à-dire effectuent la transcription. Les enzymes de la classe ARN polymérase sont très importantes pour le fonctionnement de la cellule, c'est pourquoi on les retrouve dans tous les organismes et dans de nombreux virus. Chimiquement, les ARN polymérases sont des nucléotidyl transférases qui polymérisent les ribonucléotides à l'extrémité 3" de la chaîne d'ARN.
Un promoteur est une séquence de nucléotides d'ADN reconnue par l'ARN polymérase comme point de départ d'une transcription spécifique ou significative. Chez les procaryotes, le promoteur comprend un certain nombre de motifs importants pour sa reconnaissance par l'ARN polymérase, en particulier les séquences dites -10 et -35. Le promoteur est asymétrique, ce qui permet à l'ARN polymérase de commencer la transcription dans le bon sens et indique lequel des deux brins d'ADN servira de matrice pour la synthèse de l'ARN.
La région promotrice au sein de l'opéron peut chevaucher partiellement ou ne pas chevaucher du tout la région opératrice du cistron (gène).
Le promoteur sous lequel se situe la région d'ADN codant l'ARN joue un rôle déterminant dans l'intensité de l'expression de ce gène dans chaque type cellulaire spécifique. L'activation du promoteur est déterminée par la présence d'un ensemble différent de facteurs de transcription dans chaque type cellulaire.
Le terminateur est une séquence de nucléotides d'ADN reconnue par l'ARN polymérase comme signal pour arrêter la synthèse de la molécule d'ARN et la dissociation du complexe de transcription.
Souvent, ces séquences se terminent par une chaîne de nucléotides thymine (dans les transcrits - uridine), qui est précédée d'une région contenant des séquences internes mutuellement complémentaires dans des orientations opposées (structures en épingle à cheveux). La région en épingle à cheveux est enrichie de paires GC, conférant à cette structure une plus grande stabilité.
78. Différences dans le processus de transcription dans différentes cellules. Structure mosaïque des gènes. Synthèse de l'ARN hétéronucléaire, sa conversion en ARN messager. Épissage. Traitement.
Certains virus (comme le VIH, responsable du SIDA) ont la capacité de transcrire l'ARN en ADN. Le VIH possède un génome à ARN intégré à l'ADN. En conséquence, l’ADN du virus peut être combiné avec le génome de la cellule hôte. La principale enzyme responsable de la synthèse de l’ADN à partir de l’ARN est appelée reversease. L’une des fonctions de la reversetase est de créer de l’ADN complémentaire (ADNc) à partir du génome viral. L'enzyme ribonucléase H associée clive l'ARN et la reversease synthétise l'ADNc à partir de la double hélice d'ADN. L'ADNc est intégré dans le génome de la cellule hôte par l'intégrase. Le résultat est la synthèse de protéines virales par la cellule hôte, qui forment de nouveaux virus. Dans le cas du VIH, l'apoptose (mort cellulaire) des lymphocytes T est également programmée. Dans d’autres cas, la cellule peut rester un propagateur de virus.
Certaines cellules eucaryotes contiennent l'enzyme télomérase, qui présente également une activité de transcription inverse. Avec son aide, des séquences répétitives dans l'ADN sont synthétisées. La télomérase est souvent activée dans les cellules cancéreuses pour dupliquer indéfiniment le génome sans perdre la séquence d'ADN codant pour la protéine.
Une caractéristique distinctive de la structure de nombreux gènes eucaryotes est la structure en mosaïque (discontinue) de la partie sémantique des gènes.
Cette différence est associée à l'alternance de régions sémantiques qui portent des informations sur la séquence d'acides aminés dans la protéine - les exons, et de sections de séquences non codantes nettement plus longues que les exons - les introns. En conséquence, la longueur totale du gène s’avère être environ 5 à 7 fois plus longue que ce à quoi on pourrait s’attendre.
Au début du gène (avant sa partie sémantique) se trouvent des zones qui assurent une bonne régulation du gène. Ils sont divisés en deux groupes : non spécifiques (les mêmes pour tous les gènes) et spécifiques (caractéristiques uniquement pour un gène donné). Les régions régulatrices non spécifiques chez les eucaryotes sont appelées « TATA-BOX ». Ils alternent entre la thymine et l'adénine. Cette région se situe 30 nucléotides à gauche du début de lecture du gène. Il a été établi que l'ARN polymérase repose sur l'ADN de telle manière que sa partie reconnaissance ferme la TATA BOX, et que le centre actif de l'ARN polymérase apparaît au-dessus du premier nucléotide à lire. Vient ensuite la région promotrice, constituée d'un site de reconnaissance, d'un site de liaison et d'un site d'initiation de la transcription (A ou D). La combinaison de nucléotides dans le promoteur est telle que si le cadre de lecture est mal défini, une séquence caractéristique des codons d'arrêt est obtenue et la transcription s'arrête. La région promotrice est suivie d’un palindrome (« flip ») ou d’une répétition inversée. Il s'agit d'une séquence d'ADN double brin qui se lit de la même manière dans les deux sens (« Cosaque » - de droite à gauche et de gauche à droite se lit de la même manière). Parfois, une telle séquence est appelée « région doublement symétrique », où l’axe de symétrie est le point central par rapport auquel la séquence reste la même. Une propriété importante des palindromes est la capacité à former des structures en épingle à cheveux dans l’ARN ou des structures croisées dans l’ADN. Si le cadre de lecture est mal installé, le palindrome de l'ADN se transforme en une « croix », ce qui rend impossible la progression de l'enzyme. Parfois, les palindromes peuvent être situés à la fin d'un gène après sa partie sémantique et, en outre, formant une « croix », ils arrêteront la synthèse de l'ARNm. La partie sémantique du gène, comme nous l'avons écrit ci-dessus, consiste en une alternance de séquences intron-exon. La partie structurelle du gène contient un promoteur et un palindrome.
Récemment, des inclusions de régulations génétiques spécifiques – des activateurs – ont été identifiées. Ils sont situés devant le gène à une distance de centaines et de milliers de paires de nucléotides. Les eucaryotes possèdent des protéines régulatrices spéciales qui reconnaissent l'amplificateur et s'y attachent. De cette façon, le gène est activé. Le génome est formé non seulement de gènes nucléaires, mais également de gènes cytoplasmiques. Dans le génome, les gènes remplissent différentes fonctions. Selon les fonctions qu'ils remplissent, les gènes sont divisés en gènes obligatoires ; ils codent pour des fonctions vitales dans des cellules de tous types (parfois appelés gènes « de ménage »), des gènes de structure, des gènes régulateurs et des gènes de ponctuation. Alors que les ARNm procaryotes (bactériens et archéens), à de rares exceptions près, sont immédiatement prêts à être traduits et ne nécessitent pas de traitement particulier, les pré-ARNm eucaryotes subissent des modifications intensives. Ainsi, simultanément à la transcription, une « édition » (épissage) se produit sur la partie déjà synthétisée de l’ARNm. Au cours du processus d'épissage, les séquences non codantes pour les protéines - les introns - sont retirées du pré-ARNm, un nucléotide modifié spécial (capuchon) est ajouté à l'extrémité 5" de la molécule, et plusieurs adénines, ce qu'on appelle la queue polyadénine, sont ajoutés à l'extrémité 3". La coiffe est reconnue par des facteurs d'initiation, des protéines responsables de la fixation à l'ARNm du ribosome, et la queue polyadénine se lie à une protéine spéciale, PAB. Généralement, ces changements post-transcriptionnels dans l’ARNm eucaryote sont appelés « traitement de l’ARNm ». La polyadénylation est nécessaire au transport de la plupart des ARNm dans le cytoplasme et protège les molécules d'ARNm d'une dégradation rapide (augmente leur demi-vie). Les molécules d'ARNm dépourvues d'une région poly-A (par exemple virales) sont rapidement détruites dans le cytoplasme des cellules eucaryotes par les ribonucléases.
L'épissage (de l'anglais splice - épisser ou coller les extrémités de quelque chose) est le processus consistant à découper certaines séquences nucléotidiques des molécules d'ARN et à joindre les séquences qui restent dans la molécule « mature » pendant le traitement de l'ARN. Ce processus se produit le plus souvent lors de la maturation de l'ARN messager (ARNm) chez les eucaryotes, au cours de laquelle, par des réactions biochimiques impliquant l'ARN et les protéines, les sections de l'ARNm qui ne codent pas pour une protéine (introns) sont éliminées et les sections qui codent pour l'acide aminé. séquence acide - les exons sont connectés les uns aux autres. Ainsi, le pré-ARNm immature est converti en ARNm mature, à partir duquel les protéines cellulaires sont lues (traduites). La plupart des gènes codant pour les protéines procaryotes n'ont pas d'introns, l'épissage pré-ARNm y est donc rare. L'épissage des ARN de transfert (ARNt) et d'autres ARN non codants se produit également chez les représentants des eucaryotes, des bactéries et des archées.
Traitement de l'ARN
Entre transcription et traduction, la molécule d'ARNm subit une série de changements séquentiels qui assurent la maturation de la matrice fonctionnelle pour la synthèse de la chaîne polypeptidique. Un capuchon est attaché à l’extrémité 5΄ et une queue poly-A est attachée à l’extrémité 3΄, ce qui augmente la durée de vie de l’ARNm. Avec l'avènement de la transformation dans la cellule eucaryote, il est devenu possible de combiner des exons de gènes pour obtenir une plus grande variété de protéines codées par une seule séquence de nucléotides d'ADN - épissage alternatif.
Pour réaliser une transcription correcte, deux types d’éléments régulateurs sont nécessaires. Les éléments réglementaires du premier type sont appelés cis-régulateurs. Ce sont des séquences d'ADN spécifiques sur un chromosome donné. Cis -les régulateurs n'agissent que sur les gènes proches. Le deuxième type est appelé trans-régulateurs. Ce sont des molécules solubles (notamment des protéines et de l’ARN) produites par un gène et qui interagissent avec d’autres gènes sur le même chromosome ou sur d’autres chromosomes. Si nous nous tournons vers l'induction génique dans lac -opéronE., coli Ce sont des molécules solubles (notamment des protéines et de l’ARN) produites par un gène et qui interagissent avec d’autres gènes sur le même chromosome ou sur d’autres chromosomes. Si nous nous tournons vers l'induction génique dans on peut alors rappeler que le gène répresseur produit une protéine répresseur qui interagit avec la séquence opérateur des gènes -opéron. Dans ce cas, l'opérateur est Ce sont des molécules solubles (notamment des protéines et de l’ARN) produites par un gène et qui interagissent avec d’autres gènes sur le même chromosome ou sur d’autres chromosomes. Si nous nous tournons vers l'induction génique dans- cis -un élément de régulation, puisqu'il ne contrôle que opéron de son propre chromosome. -opéron.(Une séquence opérateur mutante sur un autre chromosome peut ou non attacher une protéine répresseur.) Une protéine répresseur, en revanche, est
transe -opéron.-régulateur. puisqu'il est produit par un chromosome et se lie à -opérateur régulateur sur un autre chromosome (Fig. 12.5). généralement situé immédiatement avant le site où il commence
Gilbert S. Biologie du développement : en 3 volumes T. 2 : Trad. De l'anglais – M. : Mir, 1994. – 235 p.
142 _______________ CHAPITRE 12 _____________________________________________________________________________
|
Riz. 12.5. Schéma de régulation différentielle des gènes dans E. E.; montré -opéron.- Et -un élément de régulation, puisqu'il ne contrôle que-des éléments réglementaires. Dans les cellules de type sauvage, l'état inductible est caractérisé par le fait que l'ARN de la β-galactosidase n'est transcrit que lorsque le lactose est présent. En l'absence de lactose, la protéine répresseur (R) codée par le gène je, rejoint le site Internet de l’opérateur ( Ô), inhibant ainsi la transcription par l'ARN polymérase du promoteur ( p). E. Si le lactose est présent, il se lie à la protéine répresseur, ce qui fait que le répresseur ne peut pas se lier à l'ADN et que la transcription se poursuit. Le caractère soluble de ce répresseur a été montré dans des expériences sur des mutants. Coli je – Lorsque les cellules bactériennes haploïdes porteuses du gène je, devient partiellement diploïde avec le gène je + type sauvage ( -un élément de régulation, puisqu'il ne contrôle que), un répresseur de type sauvage est synthétisé, capable de rendre inductible le gène original de la ß-galactosidase. -opéron. Cette protéine répresseur est |
|
|
|
-élément réglementaire. Les séquences promoteur et opérateur sont |
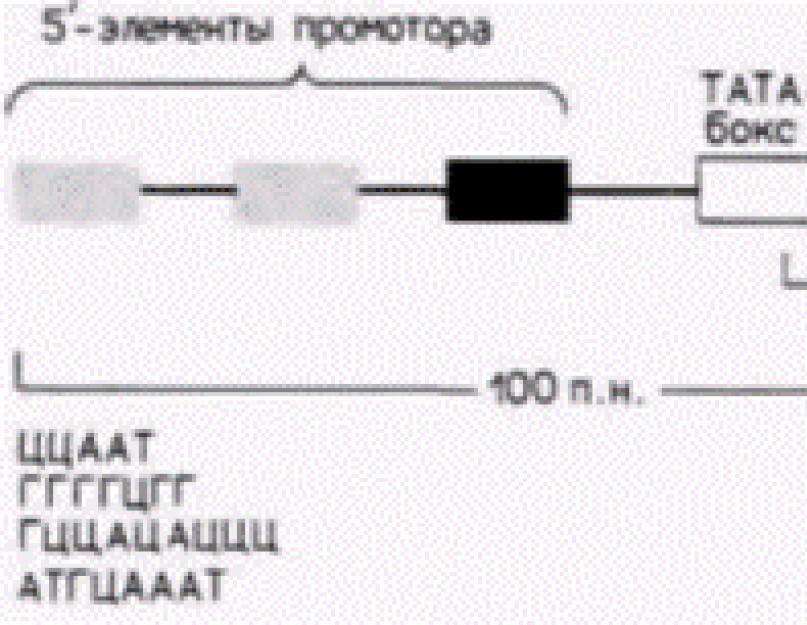
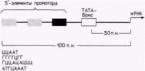
-des éléments réglementaires. -opéron. Riz. 12.6. Un promoteur typique pour un gène codant pour une protéine eucaryote. Le gène présenté contient une boîte TATA et trois éléments promoteurs 5". Des exemples de ces éléments 5' sont présentés dans la partie inférieure de la figure. (D'après Maniatis et al., 1987.)
transcription et mesurent environ 100 paires de bases. La région promotrice est nécessaire à la liaison de l’ARN polymérase II et à l’initiation précise de la transcription. L'amplificateur active l'utilisation du promoteur en contrôlant l'efficacité et le taux de transcription de ce promoteur particulier. Les Enhancers n'activent que ceux qui se trouvent à l'intérieur-position des promoteurs (c'est-à-dire des promoteurs sur le même chromosome), mais ils peuvent fonctionner sur de longues distances. De plus, ils peuvent être localisés non seulement du côté 5" du gène, mais également sur un autre brin d'ADN (Maniatis et al., 1987). Les promoteurs des gènes qui transcrivent des quantités relativement importantes d’ARNm ont des structures similaires. Ils contiennent la séquence AΤA (parfois appelée boîte TATA ou, se trouvant encore plus loin du côté 5". L'élément promoteur en amont est généralement une variation de la séquence CAAT, mais d'autres éléments promoteurs ont été identifiés (Grosschedl et Birnstiel, 1980 ; McKnight et Tjian, 1986) (Fig. 12.6).
Pour la première fois, le promoteur du gène β-globine a été étudié dans le cadre d’expériences visant à tester la transcription spécifique de l’ADN cloné. Les gènes clonés peuvent être transcrits correctement lorsqu'ils sont introduits dans les noyaux d'ovocytes ou de fibroblastes de grenouille ou lorsqu'ils sont incubés avec de l'ARN polymérase purifiée en présence de nucléotides surnageants (Wasylyk et al., 1980). Une fois la transcription d'un gène confirmée, des enzymes de restriction sont utilisées pour produire des divisions spécifiques dans ce gène ou dans les régions environnantes. Il est alors possible de déterminer si le gène modifié continue à être correctement transcrit. Les résultats de ces études ont montré que les 109 premières paires de bases précédant le site cap sont suffisantes pour une transcription maximale du gène de la ß-globine (Grosveld et al., 1982 ; Dierks et al., 1983).
D'autres chercheurs ont clarifié cette conclusion en clonant une région du gène de la globine de souris depuis la 106ème paire de bases en amont (du côté 5") du début de la transcription (position -106) jusqu'à la 475ème paire de bases (position +475) dans le premier exon (Myers et al., 1986). Ces clones ont été mutagénisés in vitro dans la région promotrice du gène de la globine.
Transcription est la synthèse d'ARN sur une matrice d'ADN. Chez les procaryotes, la synthèse des trois types d'ARN est catalysée par un complexe protéique complexe - l'ARN polymérase.
La synthèse de l'ARNm commence par la découverte par l'ARN polymérase d'une région spéciale dans la molécule d'ADN, qui indique l'endroit où commence la transcription - promoteur Après s'être liée au promoteur, l'ARN polymérase déroule le tour adjacent de l'hélice d'ADN. Deux brins d'ADN divergent à ce stade et sur l'un d'eux, l'enzyme synthétise l'ARNm. L'assemblage des ribonucléotides en chaîne s'effectue dans le respect de leur complémentarité avec les nucléotides de l'ADN, et également de manière antiparallèle par rapport au brin matrice d'ADN. L'ARN polymérase est capable d'assembler un polynucléotide uniquement de l'extrémité 5" à l'extrémité 3" ; un seul des deux brins d'ADN peut servir de matrice pour la transcription, à savoir celui faisant face à l'enzyme avec son extrémité 3" (3" → 5") Cette chaîne est dite codogène.
Terminateur- c'est la zone où la croissance de la chaîne d'ARN s'arrête et où elle est libérée de la matrice d'ADN. L'ARN polymérase se sépare également de l'ADN, ce qui restaure sa structure double brin.
Un fragment d'une molécule d'ADN, comprenant un promoteur, une séquence transcrite et un terminateur, forme une unité de transcription - transcription.
La régulation des opérons (c'est-à-dire la régulation au niveau transcriptionnel) est le principal mécanisme de régulation de l'activité des gènes chez les procaryotes et les bactériophages.
Opéron - une tranche de matériel génétique dont la transcription s'effectue par molécule d'ARN sous le contrôle d'une protéine répresseur.
Un opéron est constitué de gènes structurels étroitement liés codant pour des protéines (enzymes) qui réalisent les étapes successives de la biosynthèse d'un métabolite. Chaque opéron contient : un promoteur, un opérateur et un terminateur.
Opérateur- séquence nucléotidique qui se lie protéine répresseur et régulant négativement transcription voisin gène. L'opérateur est situé entre le promoteur et les gènes de structure. Il peut être associé à une protéine spéciale - un répresseur, qui empêche l'ARN polymérase de se déplacer le long de la chaîne d'ADN et empêche la synthèse des enzymes. Ainsi, les gènes peuvent être activés et désactivés en fonction de la présence de protéines répresseurs correspondantes dans la cellule.
Répresseur- une protéine régulatrice qui supprime la transcription des gènes de l'opéron qu'elle régule suite à sa liaison à l'opérateur (site régulateur de l'opéron). Ceci conduit à l'arrêt de la synthèse de l'ARNm correspondant et, par conséquent, des enzymes codées par l'opéron. Le répresseur est synthétisé sous le contrôle du régulateur génique à raison de 10 à 20 molécules par cellule sous forme de formes actives, c'est-à-dire capables de se lier directement à l'opérateur, ou de formes inactives. La formation d'un répresseur actif est caractéristique des enzymes inductibles, dont la synthèse ne commence que lorsque des substances spécifiques de faible poids moléculaire - inducteurs - pénètrent dans la cellule. . Inducteur- une petite molécule effectrice qui se lie à une protéine régulatrice, ou à un facteur physique (lumière, température) qui stimule l'expression de gènes inactifs.
Séquence promoteur-régulatrice de la région 5' du gène, qui détermine le site d'attachement de l'ARN polymérase à l'ADN. Le promoteur contient deux séquences qui jouent un rôle important dans l'initiation : la boîte TATA et le domaine CCAAT. Le promoteur indique à l’ARN polymérase où commencer la synthèse.
102 questions. Expliquer les concepts : transcription et transcriptome.
Transcription– une molécule d’ARN formée à la suite de la transcription d’une section d’ADN
Transcription-la totalité de tous les transcrits synthétisés par une cellule, comprend l'ARNm et l'ARN non codant
Question : Quelles molécules sont synthétisées par l’ARN polymérase I ?
Synthèse 5,8srRNA, 18srRNA, 28srRNA
responsable de la synthèse des gros ARNr, il est localisé dans le nucléole.
Question : Quelles molécules sont synthétisées par l’ARN polymérase II ?
Synthétise l'ARNm, le snRNA(faiblement nucléaire), le miARN, le miARN(faiblement interférant),
ARNm, il est localisé dans le cytoplasme
Question : Quelles molécules sont synthétisées par l'ARN polymérase III.
Synthèse de l'ARN5s, de l'ARNt, d'une partie du snARN et des petits ARNr
Question : À quoi se résume le mécanisme de transcription (synthèse d’ARN sur une matrice d’ADN).
À la suite de la transcription, un transcriptiARN primaire est formé.
Question 107 : Qu'appelle-t-on traitement de l'ARN ? Nommez de quels processus individuels il s’agit.
Le processus de maturation de l’ARNm est appelé traitement. Coiffage, polyadénylation, épissage
Question : Quelle est la réaction d’épissage des transcrits d’ARN ? Le rôle du splicingosome dans ce processus.
Épissage- Il s'agit de la coupure des introns d'une molécule d'ARNm et de l'assemblage des exons à l'aide d'enzymes ligase.
Le processus macromoléculaire établi implique un grand nombre de macromolécules. La structure macromoléculaire est réalisée (épissage->petit ARN nucléaire). Les snRNA reconnaissent la frontière entre les exons et les introns et s’y lient.
Question 109 : Quand se produit le coiffage de l'extrémité 5' de l'ARN chez les eucaryotes et qu'est-ce que cela signifie ? Transcriptions dont les ARN polymérases sont soumises à un plafonnement.
Immédiatement après la transcription. Un résidu guanosine méthylé est attaché à l'extrémité 5" de l'ARNm ; cette structure est appelée capuchon. Le capuchon facilite la liaison de l'ARNm au ribosome dans le cytoplasme.
Uniquement les transcrits de l'ARN polymérase II
Question : Énumérez toutes les dispositions concernant l'importance du plafonnement de l'ARNm.
Assure l’efficacité de la transcription ultérieure.
Protège le transcrit de la dégradation par les 5'-exonucléases (liaisons 5'-5')
Une façon de faire avancer le processus : stimule la 3'-polyadénylation et l'épissage.
Requis pour l'exportation de l'ARNm du noyau
Assure la liaison de l'ARNm au ribosome dans le cytoplasme
111.Qu'est-ce que la polyadénylation de l'extrémité 3' de l'ARNm. Quel rôle joue ce processus ? Transcriptions dont les ARN polymérases subissent ce processus.
Polyadénylation– de 100 à 200 nucléotides adényles sont ajoutés à l'extrémité 3" de la molécule d'ARNm. Une région poly-A est formée. Cette région stabilise la molécule d'ARNm et favorise sa libération du noyau vers le cytoplasme.
Transcriptions d'ARN-polymérase II
Qu’est-ce que l’épissage alternatif ? Quelle signification biologique cette forme d’épissage a-t-elle ?
l'épissage alternatif est une forme d'épissage dans laquelle la jonction des exons pendant la maturation de l'ARNm se produit dans différentes combinaisons. Dans ce cas, l’ordre des exons n’est pas violé. L'épissage garantit que les gènes codent pour différentes protéines, ce qui constitue un mécanisme de diversité protéique chez les eucaryotes (plus de 70 % des gènes humains subissent un épissage alternatif).